GPUより安く、かつ高速な推論を実現。Amazon EC2 Inf1・Inf2インスタンスの徹底活用法
本記事は、株式会社ゼンプロダクツの清原弘貴さんによる寄稿です。
Shodoの開発・運用に携わる中で得た実践的な知見をもとに、自社でAIモデルをホスティングする際の技術的な工夫を紹介します。特に、AWS Inferentia(Inf1、Inf2インスタンス)を活用して、安価かつ高性能な推論基盤を構築する方法を解説します。
はじめに
AI校正のShodoでは、自社のAIモデルを動かしてお客様の文章を校正しています。ですがAIを自分でホストするのは簡単ではありませんし、他のミドルウェア等と比べると費用もかかるものです。この記事では自身のAIモデルを、より安価で手軽にホストする方法を紹介します。BERTとLlamaベースの日本語モデルを例にしますので、実務で活用しやすいコード例をお見せできると思います。
AWS Inferentia(Inf1、Inf2)を使う利点
AWS Inferentiaを使うと高性能な推論サーバーを手頃な価格で利用できるのが魅力です。ここではAmazon EC2(以下、EC2)やAmazon SageMaker(以下、SageMaker)で使えるInf1とInf2というインスタンスに注目します。Inf1、Inf2インスタンスにはAWS独自の推論チップが搭載されており、GPUで行うような画像分析や自然言語処理などの推論タスクを実行できます。
「独自のチップ」というと手間が多かったり、ベンダーロックインになったりする心配がありますよね。ですがご安心ください。Inf1、Inf2に使うモデルは通常のBERTやLLMで問題なく、これをInf1、Inf2向けにコンパイルして、それをホスティングするだけです。この記事ではInf1、Inf2インスタンス向けのAIモデルの導入方法や対応が必要な点を紹介します。
料金の比較
利用を検討するにはまず料金が気になるかと思います。EC2での料金は2025年3月時点の東京リージョンでは以下となっています。
- inf2.xlarge: USD 1.1373
- inf1.xlarge: USD 0.308
- g6.xlarge: USD 1.1672
- g5.xlarge: USD 1.459
- g5g.xlarge: USD 0.5669
引用元:https://aws.amazon.com/jp/ec2/pricing/on-demand/
料金的には inf1.xlarge が他と比較して最も安い選択肢となっています。この場合でも1ヶ月(30.5日)稼働すると考えると、225.4ドル(1ドル150円として33,818円ほど)かかります。3万円以上となると他のインフラストラクチャーと比べると高価な部類に入りますので、自社のBERT等のAIモデルを稼働するとなるとInf1が選択肢に入ってくるでしょう。
速度を比較するとInf2(最新のAWS Inferentia)が最もコスパ良くレスポンスも早い選択肢になるでしょう。実際にShodoで稼働させているBERT系列のモデルでは、Inf1からInf2に変えることでレスポンスタイムが半分になりました。以下のグラフはShodoの推論サーバーのレスポンスタイムを記録したもので、サーバー移行の前後で半分の時間になっていることが分かります。
Inf2移行でレスポンスタイムが半分に
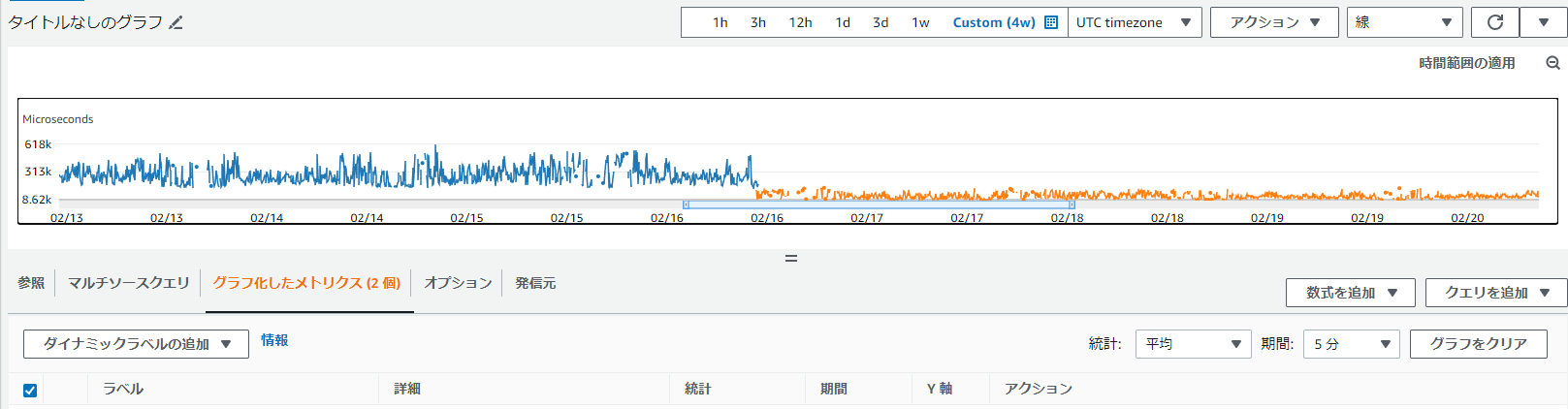
また、Inf2はLlamaにも正式対応しておりますので、モデルサイズの大きいLLMを稼働する場合もInf2が必要となります。
モデルをコンパイルする方法
Inf1、Inf2向けにモデルをコンパイルして利用する方法を紹介します。それほど複雑ではありませんし、コンパイル作業はローカルのPCでも可能ですのでご安心ください。ここでは日本語のRoBERTaのモデルで説明します(他のモデルでも基本的な手順は同じです)。
Inf2では torch-neuronx というライブラリを使ってモデルをコンパイルします。このライブラリはAWS Neuronのパッケージインデックスを指定してインストールしましょう。今回はPython 3.10、Torch 1.13のUbuntu環境で以下のインストールを確認しております。ローカルでコンパイルするためにTorch 1.13の環境を紹介していますが、コンパイル済みのモデル自体は2系でも動作します。
$ pip install --extra-index-url https://pip.repos.neuron.amazonaws.com torch==1.13.1 torch-neuronx transformers==4.49.0
今回扱うモデルで必要なパッケージもインストールします。この点は使いたいモデルの仕様に従ってください。
$ pip install sentencepiece
以下のようなプログラムを用意し、実行すればモデルをコンパイルできます。
import torch_neuronx
from transformers import AutoModelForMaskedLM, AutoTokenizer
# モデルの読み込み
model_name = "rinna/japanese-roberta-base"
tokenizer = AutoTokenizer.from_pretrained(model_name)
model = AutoModelForMaskedLM.from_pretrained(model_name)
# traceに使うダミー文の用意(モデルの仕様上[CLS]を足す)
dummy = "[CLS]これはダミーの文です。"
inputs = tokenizer(
dummy,
max_length=128,
padding="max_length",
return_tensors="pt",
)
# モデルのコンパイル
input_ids = inputs["input_ids"]
attention_mask = inputs["attention_mask"]
# モデルに対するダミーの入力を位置引数として渡す(モデルの利用時もこの渡し方となる)
model_neuron = torch_neuronx.trace(model, (input_ids, attention_mask))
# モデルの保存
model_neuron.save("neuron_model.pt")
これで neuron_model.pt というInf2用のモデルがコンパイルできました。このモデルをInf2インスタンスで読み込むことで通常のモデルのように扱えます(詳しくは後述)。
Inf1の場合は、 torch_neuronx ではなく torch.neuron というライブラリを利用します。インストールのうえ、上記のプログラムのうち torch_neuronx となっている点を torch.neuron に置き換えるだけでOKです。ただしtorch.neuronライブラリの対応しているPyTorchのバージョンや、そのPyTorchが対応しているPythonのバージョン等に気を付けてください。
次に、コンパイル済みモデルの特徴を見ていきましょう。
コンパイル済みモデルの違い
いくつかの点で、通常のモデルとの違いがあります。
- 引数を位置引数で渡す必要がある
- 入力長を決めておく必要がある
モデルのコンパイル時にも、位置引数で input_ids と attention_mask を渡しており、入力長も128文字に固定しています。これと同様に、コンパイル済みモデルを利用する際も同じように「位置引数」、「固定長」で利用します。
通常のモデルはキーワード引数で model(input_ids=...) のように渡せるがそれができない点に注意してください。transformers のPipelineがモデルを呼びだすときもキーワード引数なのでカスタムが必要となります。
EC2でコンパイルしたモデルを使う方法
EC2でInf2、Inf1インスタンスを使い、コンパイルしたNeuronモデルを動かしてみましょう。このとき、EC2インスタンスの起動時にはDeep Learning AMI Neuronを使う必要があります。起動時にAMIの検索欄にこの名前を入れて、選択してください(今回は Deep Learning AMI Neuron (Ubuntu 22.04) を利用)。このAMIを使うと事前に環境構築が済んだPythonの仮想環境を使えます。
Neuron AMIの場合、仮想環境は /opt 以下に置かれているのでこれを有効化しましょう。さまざまなバージョンに対応した仮想環境がありますが、ここではInf2(Neuronx)でTorch、Transformersを使う場合の仮想環境を使います。
$ source /opt/aws_neuronx_venv_pytorch_2_5_transformers/bin/activate
この時点でsentencepieceなど必要なライブラリをインストールしてください。
次にコンパイル済みのモデルを転送したうえで、以下のようにプログラムを実行するだけです。以下のコードの outputs には通常のモデルで実行したのと同じように推論の結果が格納されます。
import torch
import torch_neuronx
from transformers import AutoTokenizer
tokenizer = AutoTokenizer.from_pretrained("rinna/japanese-roberta-base")
model = torch.jit.load("neuron_model.pt")
inputs = tokenizer(
"[CLS]佐々木朗希、メジャーデビューの[MASK]は東京ドームで。",
max_length=128,
padding="max_length",
return_tensors="pt",
)
outputs = model(inputs["input_ids"], inputs["attention_mask"])
これでAWS Neuron(Inf2、Inf1)環境でのモデルの実行ができました。最初からPythonの仮想環境が用意されているため、インストールや特殊な環境構築の必要がありません。またコンパイル済みモデルについても通常のTransformersのモデルとほとんど同じように扱えます。
もっと簡単な方法はないの?
optimum-neuronというライブラリを利用することで、より簡単にAWS Neuronに対応したモデルのコンパイルや実行ができます。たとえばCLIツールからモデルのコンパイルが一発でできます。
$ optimum-cli export neuron \
--model rinna/japanese-roberta-base \
--batch_size 1 \
--sequence_length 128 \
neuron_model/
さらに optimum.neuron.NeuronModelForXXX (NeuronModelForMaskedLM など)というモデル用のクラスを使えば、通常のTransformersのモデルのようにコンパイルしたディレクトリ(上記の neuron_model)を読み込めます。この場合、位置引数の問題も自動的に解決してくれるため、通常のTransformersのモデルのように扱えます。
ただし注意も必要です。このライブラリはまだ開発が盛んに行われているもので、2025年3月時点で0.1.0がちょうどリリースされたばかりです。それまで0.0.28などのバージョンとしてリリースされていることを思えば安定したほうかもしれませんが、使われる際は最新の情報を確認したうえで使うことをおすすめします。比較的新しいTransformers環境を要求されたり、互換性の対応範囲も狭かったりしますが、使えると便利ですのでぜひ活用してみてください。
Inf1、Inf2はSageMakerで使うのもおすすめ
SageMakerを使えば面倒な設定をせず、簡単にモデルをデプロイし推論用のAPIを作成できます。AWS Neuron向けのコンパイル済みのモデルでも、S3上に配置したパスからデプロイが可能ですし、無停止でのモデルの更新も可能です。またTerraformもSageMakerの推論エンドポイントに対応しているので、環境の管理を行っている場合も適用できます。
この記事ではSageMakerでの利用方法を簡単に紹介します。
SageMakerで推論用スクリプトを実行する
SageMakerはモデルのディレクトリ内に code というディレクトリと code/inference.py というファイルを配置してプログラムをカスタムできます。このファイル内でSageMakerの規則に従うことでさまざまなカスタムが可能です。また code/requirements.txt を配置すれば依存ライブラリの追加もできます。
おすすめは model_fn をカスタムしてPipelineを返す方法です。他に細かいことを気にせず、 code/inference.py の model_fn 関数からTransformersのPipelineを返せば、API越しにこれを実行して結果を得られるようになるのです。
model_fn が受け取る model_dir の引数にはSageMakerの起動時に指定したTarファイルを展開したディレクトリへのパスが指定されます。ここからコンパイル済みのモデルを読み込んでPipelineとして返すだけでOKです。
コンパイルしたモデルは入力を位置引数で指定する必要があるため、そのためのカスタムを行いましょう。この例ではBERT系のモデルでトークンの分類を行う例を示しています。 inference.py は以下のようになります。
class MyNeuronPipeline(TokenClassificationPipeline):
def _forward(self, model_inputs):
# Pass params of models as positional arguments
output = self.model(model_inputs["input_ids"], model_inputs["attention_mask"])
logits = output["logits"] if isinstance(output, dict) else output[0]
return {
"logits": logits,
**model_inputs,
}
def model_fn(model_dir):
tokenizer = AutoTokenizer.from_pretrained(model_dir)
config = AutoConfig.from_pretrained(model_dir)
config.max_length = 128
model = torch.jit.load(os.path.join(model_dir, "neuron_model.pt"))
model.config = config
return MyNeuronPipeline(
model=model,
tokenizer=tokenizer,
framework="pt",
)
位置引数での指定方法に対応するため、Pipelineをカスタムしています。先述したoptimum.neuronが提供するNeuronModelForXXXを使うことでもこの問題は解決できます。
model_dir のディレクトリは .tar.gz に圧縮したものを指定することでSageMakerが自動的に展開してくれます。この例ではたとえば以下のようなディレクトリ構成になります。
- neuron_model.pt
- code
- inference.py
- requirements.txt
- 他、モデルのコンフィグやトークナイザーの設定
SageMakerのこのモデルの読み込み処理や、inference.py のカスタムについてはこのツールキットの挙動に従います。参考にすることでやりたいことにあわせたカスタムや拡張が可能です。
https://github.com/aws/sagemaker-inference-toolkit
SageMakerで他のカスタムを行う方法
他にも推論にはさまざまな環境構築が必要な場合があります。Pythonのライブラリを追加したり、サーバー環境にパッケージをインストールしたりが必要になるでしょう。
Pythonのライブラリを追加したい場合は inference.py と同じように code/requirements.txt を配置するだけで済みます。
sentencepiece
たとえばInf1をSageMakerで利用する場合、デフォルトで提供されている環境が少し古いバージョンのライブラリを使うため、 requirements.txt に新しいライブラリを指定して更新が可能です。Pythonのバージョンやその他のライブラリとの互換性には注意してください。
OSレベルで追加パッケージのインストールが必要な場合は、Dockerコンテナーのカスタムが有効です。SageMakerが利用しているコンテナーをもとに自分でビルドし、ECRにプッシュして指定が可能になります。たとえば形態素解析にJumanなどが使われている場合などはこの方法が必要になります。
SageMakerの対応Dockerコンテナーはこちらに記載されているので、このDockerイメージを拡張したイメージをECRのプライベートリポジトリに配置しましょう。SageMakerの起動時に指定して使えるようになります。
https://github.com/aws/deep-learning-containers/blob/master/available_images.md/
この記事ではSageMakerでの細かなカスタム方法やTerraformでの設定方法は割愛しますが、気軽にAPIのエンドポイントとしてモデルを安全にデプロイできるのでおすすめします。
おわりに
EC2でInf1、Inf2インスタンスを利用する方法を紹介しました。Inf1、Inf2(AWS Neuron)は安価に高性能なAIサーバーが手に入るためおすすめします。AIのモデルについてもコンパイルするだけで利用できるためベンダーロックインの心配なども少ないのが利点です。ぜひInf1、Inf2を活用して自前のAIをAWSで稼働させましょう。
筆者プロフィール

清原弘貴
X: https://x.com/hirokiky/
GitHub: https://github.com/hirokiky/
株式会社ゼンプロダクツ代表取締役
DjangoCongress JP、django-ja代表










